Notícias LNCC
LNCC e UFRJ identificam nova linhagem do SARS-Cov-2 no Rio de Janeiro
Publicado em: 22/12/2020,14:22 Última atualização em: 28/12/2020, 10:32
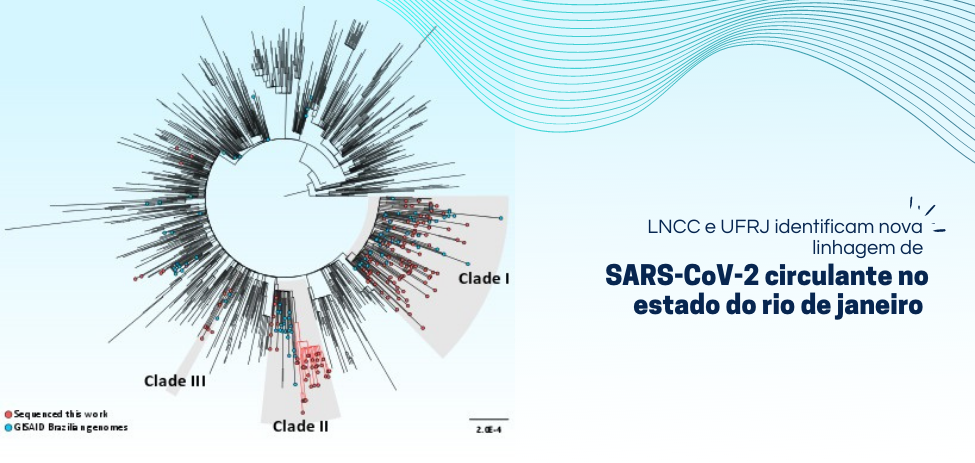
Segundo análise, a nova linhagem foi identificada em 38 dos 180 genomas sequenciados.
O Laboratório de Bioinformática - Labinfo, do Laboratório Nacional de Computação Científica - LNCC/MCTI (unidade de pesquisa do Ministério da Ciência, Tecnologia e Inovações), identificou cinco mutações, caracterizando uma possível nova linhagem originária do B.1.1.28 do novo Coronavírus. A análise, conduzida em colaboração com o Laboratório de Virologia Molecular da Universidade Federal do Rio de Janeiro - UFRJ, as Secretarias de Saúde de Maricá e do Rio de Janeiro e o Laboratório Central de Saúde Pública Noel Nutels sequenciou 180 genomas do SARS-CoV-2, provenientes de amostras do Estado do Rio de Janeiro.
A B.1.1.28 já circulava no Brasil no início do ano. As mutações são no C100U, C28253U, G28628U, G28975U e C29754U. Além dessas cinco, a mutação G23012A (E484K), no domínio de ligação ao receptor da proteína Spike, está amplamente espalhado nos genomas dessa linhagem. E484K foi anteriormente associada ao escape de anticorpos neutralizantes contra SARS-CoV-2.
A pesquisadora Ana Tereza Ribeiro de Vasconcelos, que é geneticista e bioinformata com doutorado em Genética pela UFRJ e que tem experiência na área de Bioinformática e de Biologia Computacional, atuando principalmente na integração de dados das várias camadas multi-ômicas através da computação de alto desempenho, além de responsável pelo Labinfo do LNCC, explica que de acordo com as análises filogenéticas, o surgimento desta nova linhagem ocorreu em julho de 2020 e foi identificada, principalmente, no Rio de Janeiro, em Cabo Frio, Niterói e Duque de Caxias na Baixada Fluminense.
Segundo Ana Tereza não existe indicação que essa linhagem seja mais transmissível ou que possa interferir na efetividade das vacinas que estão sendo desenvolvidas. Entretanto, ela ressalta a importância de estudos contínuos de vigilância genômica para análise da dispersão dessa nova linhagem e na identificação de novas variantes do SARS-CoV-2 no estado do Rio de Janeiro e no Brasil.
As análises indicam que a linhagem B.1.1.28 aparece como emergente, sendo identificada em 38 dos 180 genomas sequenciados. Por outro lado, os pesquisadores apontam que a linhagem B.1.1.33 está em declínio.
O trabalho foi financiado pela Faperj, Ministério da Ciência, Tecnologia e Inovações submetido em 20 de dezembro ao MedRxiv. Confira o artigo publicado: https://www.medrxiv.org/content/10.1101/2020.12.23.20248598v1